![]()
![]()
![]()
Use LEFT and RIGHT arrow keys to navigate between flashcards;
Use UP and DOWN arrow keys to flip the card;
H to show hint;
A reads text to speech;
30 Cards in this Set
- Front
- Back
|
verschienden Symbiosearten |
Mutualismus= beide Spezies profitieren KOmmensalismus = eine Art profitiert und bei einer passiert nichts Parasitismus= eine Art profitiert und einer wird geschadet |
|
|
Mikrobielle Gilde |
metabolische "verwandte" mikrobielle Populationen die nicht tatsächlich verwandt sein müssen
Mehrere Gilden in einer mikrobiellen Gemeinschaft die mit makroorganismen und abiotischen Faktoren interagieren bilden ein Ökosystem |
|
|
Nische |
+ Habitat das von einer Gilde genutzt wird +ist von Umwelteinflüssen begründet + liefert Nährstoffe und Bedingungen die zu Wachstum führen |
|
|
Microenvironment |
die direkt umgebende Umwelt eines Mikroorganismus und einer Gruppe davon
|
|
|
Most-probable Numer (MPN) |
1ml Flüssigkeit wird in 9ml verdünnt, davon wieder 1ml in 9ml usw. bis es kein Wachstum mehr gibt |
|
|
Warum wird genau das 16S rRNA Molekül verwendet? |
+ funktionell konstant + gibt es in allen Domänen des Lebens + relativ großer Informationsgehalt + verschiedenste Ausmaße an Konservierung + fast kein horizontal Gentransfer |
|
|
rRNA Approach für Reinkulturen |
Reinkultur => 16S rRNA amplification => 16S rRNA Sequenzierung => Phylogenetische Identifikation
Moderner Zugang: statt molekularem Klonen werden NGSs eingesetzt |
|
|
PCR-Schritte |
1. Denaturierung: 1min bei 94°C 2. Akühlen 45s bei 54°C wodurch die forward und reverse primer binden können 3. Verlängerung: 2min bei 72°C durch das Anbinden von dNTP's durch die Taq-Pol |
|
|
Sanger Sequenzierung |
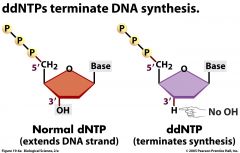
Kettenabbruch Methode: 4 Bahnen werden gemacht jewils mit einem gefärbten Nukleotid welches ein ddNTP ist
|
|
|
paralogour Rooting |
Paraloge Gene sind welche deren gemeinsamer Vorfahre eine Genverdopplung passiert ist und
Ortholog sind Gene deren Urahn einen Artgenese durchlebt hat.
Bsp. für Gene die sich routen lassen: Elongationsfaktor EF-Tu und EF-G |
|
|
Arten lateralen Gentransfers |
+ Conjugation: durch Ausbildung einer Plasmabrücke + Transduktion: durch Gentransfer eines Phagen + Transformation: freie Übertragung von Genen |
|
|
molecular cloning |
= Vervielfältigung von definierten DNA Sequenzen in vivo (z.B. in E.Coli)
1. PCR Producte (z.B. 16S-rRNA Gene von verschiedenen Organismen werden in Vektoren (z.b. Plasmide) ligiert welche in E. Coli transformiert werden 2. durch zugabe von Antibiotika und einer Antibiotikaresisdenz auf den Plasmiden werden die Zellen ohne Plasmid aussortiert 3. die E. Coli mit den Vektoren werden ausplatiiert und beginnnen in Kolonien mit je einem bestimmten Plasmid zu wachsen |
|
|
Illumina |
1.an kurze DNA Fragmente werden an jeder Seite Adapter ligiert 2. die dsDNA wird zur ssDNA und kommen auf eine Flowcell mit immobilisierten Primern 3. es kommt zur bridge amplification so lange mit Cluster des selben DNA-Abschnittes entstehen, das ganze passiert noch mit mehere Zyklen (=On.Chip PCR) 4. es werden die 4 flureszensmarkierten Basen welche asl "rerversible terminators" fungieren und es kommt zum "sequencing by synthesis" |
|
|
OTUs zur Phylabestimmung |
=Operational taxonomic unit
98,7-99% ==> Spezies 95% ==> Genus 75-80% ==> Phylum |
|
|
Wie heisst das Hormon das fett macht? |
Leptin, es hemmt das Auftreten von Hungergefühl und wird hauptsächlich von Fettzellen expremiert |
|
|
rRNA approach Biases
|
natürliche: |
|
|
Full cycle rRNA approach |
1. Umweltprobe 2. 16S rRNA Gene 3. NGS (454/Illumina) um 16S rRNA Sequenzen zu bekommen 4. Vergleichsanalysen und phylogenetische Identifikation 5. Sondendesign 6. FISH bei Umweltprobe
|
|
|
Multiple Probe Concept |
Man kann die Sonden so spezifisch designen wie man will um so auf auf verschiedene Taxonomie zu testen |
|
|
DOPE FISH |
hat 2 Fluoreszensmoleküle pro Sonde um so die -Sichtbarkeit zu erhöhen
|
|
|
CARD FISH |
= CAtalyzed Reporter Deoposition mit einer Kren-peroxidase (HRP) werden die Sonden ausgestattet; dann kommen Fluoresently-labbel tyramides welche die HRP komplett belegen und das Leuchtsignal somit um ein vielfaches höher machen
Problem: HRP ist groß und die Zellwand muss permeabilisiert werden was zur lyse führen kann |
|
|
real-Time PCR |
während der 3. PCR Phase lassen sich die amplifizieren DNAs messen durch Fluoreszenzmerkmale die auch mitamplifiziert werden und welche dann mit Reportersonden sichtbar gemacht werden: TaqMan und Molecular Beacons |
|
|
Rank abundance curve |
Core taxa (>0,1-1%) Rare taxa (<0,1%)
Dank z.b. Illuminia kann durch deep sequencing auch die rare taxa erfasst werden
Core taxa ist nicht stabil und es kann sein dass sie z.b. eine keystone spezies aus der rare taxa brauchtW |
|
|
Wie viele Mikroben leben im Darm |
100 Trillionen (10^14) Dickdarm: 10^11 - 10^12/ml
|
|
|
Wie heißen die dominanten Bakterienphyla im Darm (>98%) und wobei helfen sie? |
+ Bacteroidetes + Firmicutes +Proteobacteriia + Actinobacteria
=> Immun stimulation/modulation => Phatogenschutz => Verdauung/Versorgung mit Nährstoffen => spielen eine Rolle im Gewichtsverlust oder beim Zunehmenn |
|
|
Prime niche |
ist die Nische in der ein Organismus sicher existieren kann (min 1/organismus) |
|
|
Pyrosequenzierung |
1. Primer hybridisiert mit einem DNA template wird mit DNA POL, ATP Sulfurylase und Luciferase mit dem Substrat APS und Luciferin inkubiert 2.Elognation indem nur eine der 4 Basen zugegeben wird wodurch ein PPi abgepalten wird 3. APS+PPi wird mit Sulfurylase zu ATP 4. Luciferase macht luciferin zu oxyluciferin und mit ATP erzeugt es Licht 5. Apyrase baut die Restprodukte ab |
|
|
alpha Diversität |
die Biodiv in einer Area, community oder Ökosystem und wird als species richness ausgedürckt |
|
|
Beta Diversität |
vergleicht 2 Ökosysteme und welche Taxa einzigartig in einem sind |
|
|
die 3 menschlichen enterotypen |
+Bacteroides +Prevotella +Ruminococcus |
|
|
Mikrobielle Gruppen im DIckdarm |
=> Energy metabolism + fermentative, dentrifizierende, sulfat- reduzierende, acetogenic, methanogenische Bakterien
=> General sustrate source + pimäre host compound (mucus) + Sekundäre Konsumenten
|

