![]()
![]()
![]()
Use LEFT and RIGHT arrow keys to navigate between flashcards;
Use UP and DOWN arrow keys to flip the card;
H to show hint;
A reads text to speech;
17 Cards in this Set
- Front
- Back
|
Geben Sie die Michealis-Menten-Gleichung an. Definieren Sie alle Parameter. Was bedeuten Km und Vmax? |
*beschreibt die Abhängigkeit der Reaktionsgeschwindigkeit einer Enzymreaktion von der Konzentration des Enzyms und von der Konzentration des Substrats: v=vmax[S]/Km+[S] * v = Reaktionsgeschwindigkeit * [S] = Substratkonzentration * vmax = maximale Reaktionsgeschwindigkeit * Km = Michaelis-Konstante *Die Michaelis-Konstante Km : Sie kann experimentell gemessen werden und steht in Bezug zur Dissoziationskonstanten. Damit gibt sie Hinweise auf die Affinität des Substrates (geringe Km -Werte kennzeichnen eine hohe Affinität). Über die Michaelis-Menten-Gleichung ist Km definiert als Substratkonzentration, bei der das Enzym mit halbmaximaler Geschwindigkeit arbeitet. Km gibt damit die Substratkonzentration an, bei der die Hälfte der aktiven Zentren besetzt ist. Die Kenntnis der Michaelis-Konstanten Km eines bestimmten Enzyms ermöglicht also die Untersuchung metabolischer Reaktionen: Kennt man den Km -Wert eines Enzyms, so lässt sich für jede beliebige Substratkonzentration der Anteil der besetzten aktiven Zentren bestimmen *Die andere Konstante ist kcat, die katalytische Konstante, die die Umsatzgeschwindigkeit des Enzyms angibt. kcat wird - im Gegensatz zu Km - nicht unmittelbar, sondern indirekt über v = kcat /[E]0 bestimmt. Dies setzt die Kenntnis der molaren Menge des eingesetztes Enzyms voraus. |
|
|
Nennen Sie 2 negative + polare/ungeladene Aminosäuren. |
negative (=sauer): Aspartat, Glutamat
polar +ungeladen: Serin, Threonin |
|
|
Definieren Sie Allosterie
|
dient der Enzymregulation: Allosterisch geregelte Enzyme nehmen nach Bindung des Modulators (allosterischer Effektor/ Aktivator oder Inhibitor) an die allosterische/ regulatorische Stelle eine andere Gestalt an (=Konformitätsänderung) , was eine Änderung der strukturellen Eigenschaften und der Funktionen der aktiven Stelle des Enzyms zur Folge hat |
|
|
Definieren Sie Kooperativität |
Findet nur bei oligomeren Enzymen statt (z.B. Hämoglobin): -> gegenseitige Beeinflussung der aktiven Stellen auf den verschiedenen Untereinheiten. Binden des Substrats an die aktive Stelle einer Untereinheit fördert das Binden des Substrats an die aktiven Stellen der anderen Untereinheiten. Die besetzte Stelle erhöht die Affinität der unbesetzten Stellen. |
|
|
Was bedeutet ΔG>0? |
Änderung der freien Enthalpie (ΔG)beschreibt die Energieverhältnisse bei biochemischen Reaktionen Der ΔG-Wert einer Reaktion gibt an, in welche Richtung die Reaktionunter den gegebenen Bedingungen ablaufen kann: ΔG < 0 exergoner Vorgang. Er verläuft spontan (aber evtl. sehr langsam!). ΔG > 0 endergoner Vorgang. erfordern Energieaufwand. Die Rückreaktion wäre spontan. ΔG = 0 das System befindet sich im Gleichgewicht. |
|
|
ATP-Synthase skizzieren und beschriften
|
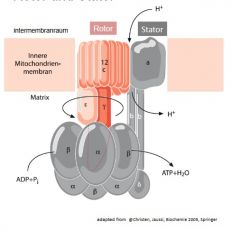
|
|
|
Zeichnen Sie ein Schema der oxidativen Phosporylierung. Vergessen Sie nicht, alle beteiligten Faktoren/ komplexe zu kennzeichnen.
|
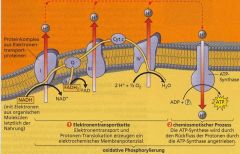
|
|
|
2 nichtkovalente Wechselwirkungen nennen und für jede davon eine Aminosäure? als Beispiel.
|
1) Wasserstoffbrückenbindung (Bsp. alle hydrophilen Aminosäuren ausser Cystein) 2) heteropolare (elektrostatische) Bindungen 3) apolare (hydrophobe) Bindungen 4) Van-der-Waals-Kräfte |
|
|
Glykosidische Bindungen zwischen Kohlenhydraten erklären |
Glycosid = Glycon (anomeres C-Atom) + Aglycon -> Abspaltung eines Wassermoleküls, Kondensationsreaktion 2 Monosaccharide (je 1 Hydroxyl (OH) -Gruppe) -> Hydroxylgruppe eines Edukts greift an C-Atom des anderen an -> Aspaltung H2O -> beide über 1 O-Atom verbunden |
|
|
die Reaktionen der Glykolyse, die ATP erzeugen nennen (Namen der Stoffe) und deren Strukturformel skizzieren. |
1. Glucose -> Glucose-6-Phosphat (ATP->ADP) 2. Fructose-6-Phosphat -> Fructose-1,6-bisphosphat (F-1,6-BP) (ATP -> ADP) Stufe 2: Spaltung von 1 mol F-1,6-BP in 2 mol Glycerinaldehyd-3-phosphat (GAP) GAP -> Glycerinsäure-1,3-BP (NAD+ -> NADH+H+) 1. Glycerinsäure-1.3-BP -> Glycerinsäure-3-P (ADP->ATP!) 2. Phosphoenolpyruvat -> Pyruvat (ADP->ATP!) |
|
|
Worin besteht der Unterschied zwischen Substrat- und Atmungskettenphophorylierung? |
Substratkettenphosphorylierung = Glycolyse oder Citratzyklus Glycolyse: 1. Phosphorylierung: Bildung von Glucose-6-phosphat (G-6-P), verbraucht ATP (Energieverlust!) (fängt Glucose in der Zelle wegen hoher Ladungsdichte) 2. Phosphorylierung: Bildung von Fructose-1,6-Bisphosphat (F-1,6-BP), verbraucht ATP (Energieverlust!) (C6-Körper wird im nächsten Schritt in 2 C3-Körper zerlegt) 1. ATP-Erzeugung durch Substratkettenphoshorylierung: -> 1,3-BPG: hohes Phosphorylgruppenübertragungspotenzial; einer seiner Phosphorylgruppe kann auf ADP übertragen 2. ATP-Erzeugung durch Substratkettenphoshorylierung: PEP: hohes Phosphorylgruppenübertragungspotenzial: einer seiner Phosphorylgruppe kann auf ADP übertragen Phosphorylierte Verbindungen mit hohem Phosphorylgruppenübertragungspotenzial übertragen in enzymkatalysierten Reaktionen eine Phosphorylgruppe auf ADP -> Gewinn Substratkettenphosphorylierung nur 2 ATP! Atmungskettenphosphorylierung = oxidative Phosphorylierung (aerob) Eine protonenmotorische Kraft, aus einem Ionengradienten (H+, selten Na+) und einem Membranpotenzial ermöglich über Rotationskatalyse die Übertragung von Phosphat auf ADP Die Geschwindigkeit der oxidativen Phosphorylierung wird durch den ATP-Bedarf bestimmt. Normalerweise fließen keine Elektronen durch die Elektronentransportkette zum Sauerstoff, wenn nicht gleichzeitig ADP zu ATP phosphoryliert wird -> Insgesamt können durch die vollständige Oxidation 1 Glucosemoleküls zu CO2 und H2O bis zu 30 Mol ATP gewonnen werden (Wirkungsgrad ca. 30 %) |
|
|
Zeichnen Sie eine Lipiddoppelschicht mit transmembranen, integralen und peripheren Proteinen. |
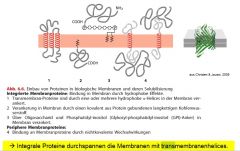
|
|
|
Die 4 Ebenen der Proteinstruktur definieren. |
1) Primärstruktur: Aminosäresequenz 2) Sekundärstruktur: H-Brücken innerhalb der Hauptkette (zwischen Peptidbindungen) -> alpha-Helices & beta-Faltblätter 3) Tertiärstruktur: dreidemsionale Konformität des Proteins (z.B. durch Ladungen der Seitenketten bedingt) 4) Quartärstruktur: hält Proteinkomplex in bestimmter geometrischer Anordnung |
|
|
Die allgemeine Gleichung für eine Säure-Base-Reaktion lautet AH + B -> A- + BH+. a) Definieren sie alle Parameter dieser Gleichung b) Wie sind der pH-Wert und der pKa-Wert definiert? |
Wasserstoffionen werden an Moleküle angehängt oder von ihnen entfernt: AH = Säure (Protonendonator) B = Base (Protonenakzeptor) A- = konjugierte Base BH+ = konjugierte Säure pH-Wert = Konzentration von Wasserstoff-Ionen in einer Lösung pKa-Wert = Maß für Stärke einer Säure ( je niedriger der pKa-Wert, umso stärker die Säure) |
|
|
Zeichnen Sie ein beliebiges Dipeptid und benennen Sie die Bindung, die die beiden Aminosäuren verknüpft. |
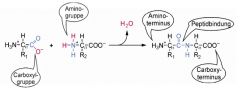
Peptidbindung Strukturmerkmal der proteinogenen Aminosäuren: zentrales C-Atom (Cα) mit vier Substituenten: Wasserstoff (H), Aminogruppe (NH2), Carboxylgruppe(COOH) sowie eine variable Seitenkette (R). |
|
|
Beschreiben Sie Aufbau und Funktion von Hämoglobin. |
oligomeres Protein aus globulären Untereinheiten: *Tetramer aus 2 alpha- und 2 beta-Untereinheiten = Zusammenlagerung von 2 Dimeren aus je 1 alpha und 1 beta bilden ein * 1 Häm-Molekül (prosthetische Gruppe) je Untereinheit (=4), welches O2-Molekül binden kann => Proteinhülle um Häm: -schützt Häm vor Oxidation -senkt Affinität von CO2 - kontrolliert Reaktivität des Häms zu O2, um reversible Bindung zu ermöglichen |
|
|
Nennen Sie jeweils 2 Aminosäuren deren Seitenketten bei neutralem pH-Wert a) negativ geladen b) positiv geladen c) unpolar/ hydrophob d) polar/ hydrophil sind. |
a) negativ (sauer): Asparaginsäure und Glutaminsäure haben Carboxylgruppe im Aminosäurerest und können ein H an Base abgeben, wodurch Seitenkette negativ geladen wird -> Aspartat, Glutamat b) positiv (basisch): Aminogruppe im Aminosäurerest -> Histidin, Lysin, Arginin c) Alanin, Valin, Leucin... (Kohlenwasserstoffreste in Seitenkette, aliphatische oder aromatische Seitenketten) d) Tyrosin, Threonin, Serin... (Hydroxyl HO-, oder Sulfhydryl HS-Gruppe in Seitenkette) |

